
Mit Computermodellierung lassen sich antivirale Medikamente künftig schneller entwickeln. (Foto von Alina Grubnyak auf Unsplash)
Medikamente gegen Viren zielgenau einzusetzen, ist eine Herausforderung. Das zeigte sich mit Sars-CoV-2 sehr deutlich. Im Zuge der Mutationen des Coronavirus verloren manche Therapien ihre Wirksamkeit. Dem möchten Forschende der Universität Tübingen entgegenwirken. Mittels Computermodellierungen sollen schnell spezifische Stoffwechselwege identifiziert werden. Sie ließen sich dann gezielt hemmen.
Das Team um Andreas Dräger, Juniorprofessor an der Universität Tübingen und Mitglied des Tübinger Exzellenzclusters „Kontrolle von Mikroorganismen zur Bekämpfung von Infektionen – CMFI“, geht davon aus, dass die Ergebnisse ihrer aktuellen Studie eine wichtige Grundlage für die schnellere Entwicklung von viralen Hemmstoffen darstellen. „Unsere Modelle könnten einen Paradigmenwechsel in der Wirkstoffentwicklung darstellen und die präklinische Phase beschleunigen“, betont Nantia Leonidou, Erstautorin der Studie und ergänzt: Die Methoden seien vollständig auf jeden Virus- und Wirtszellentyp übertragbar und auch kommerziell nutzbar.
Wie Computer gegen Viren wirken können
Mit ihrer neuartigen Analysetechnik erstellten die Wissenschaftler ein Modell und spürten damit weitere Angriffspunkte für Sars-CoV-2 auf. Konkret bedeutet das, dass das integrierte systembiologische Modell eine Corona-Infektion in bronchialen Epithelzellen simuliert. Anschließend identifiziert es wirtsbasierte Stoffwechselwege, die gehemmt werden können, um die virale Vermehrung zu unterdrücken. Kenne man die Zusammensetzung eines Virus, könne man verschiedene Szenarien durchspielen und sehen, wie sich die biochemischen Reaktionen in den Wirtszellen während einer Virusinfektion verändern, erläutert Dräger. Um eine Infektion zelltypspezifisch zu simulieren, entwickelte das Team hochwertige Software.
Seine Studie präsentiert einen neuartigen Arbeitsablauf, um robuste arzneimittelfähige Ziele gegen neu auftretende RNA-Viren vorherzusagen. Dabei werden metabolische Netzwerke und Informationen über die virale Struktur und ihre Genomsequenz verwendet.
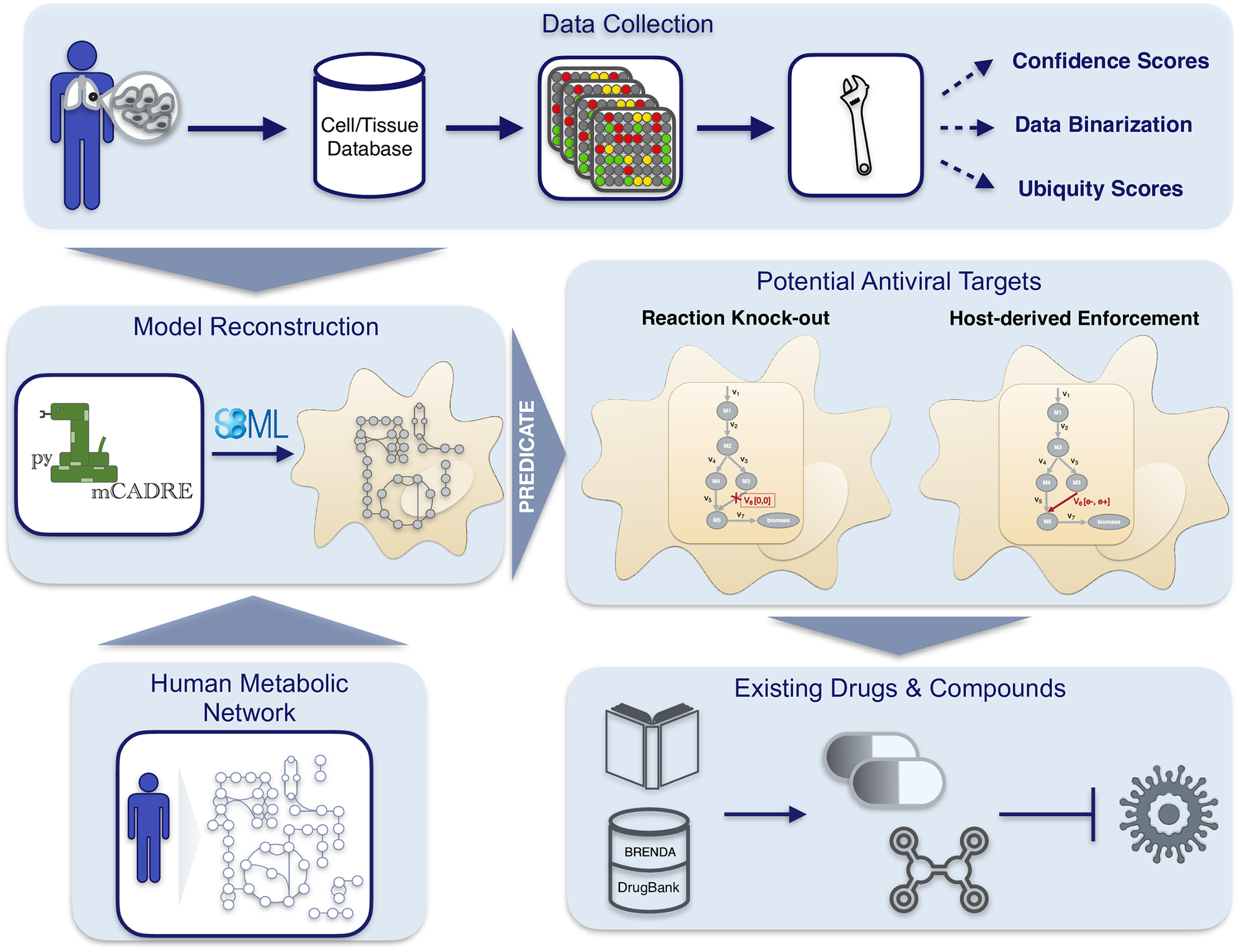
Ein neuer Arbeitsablauf sagt über Stoffwechselveränderungen in infizierten Zellen voraus, wo die Wirkstoffziele gegen Sars-CoV-2 liegen. (Fotocredit: Nantia Leonidou, Alina Renz, Reihaneh Mostolizadeh, Andreas Dräger)
Alte und neue Angriffsziele von Sars-CoV-2
In ihrer Untersuchung gelang es den Forschenden einerseits ein Angriffsziel bestätigen, das sie bereits 2021 entdeckten: Es ist das menschliche Enzym – die Guanylatkinase 1. Dieses ist für die Virusvermehrung unabdingbar und kann ausgeschaltet werden, ohne die Zelle zu schädigen.
Andererseits identifizierte das Computermodell neue Ziele mit antiviralen Wirkungen. Der vielversprechendste neue Treffer war die CTP-Synthase 1. Hemmten die Tübinger dieses Enzym, minderte es das Viruswachstum um 62 Prozent, ohne sich auf die Zellerhaltung des menschlichen Wirts auszuwirken.
Beide Zielmoleküle hängen eng mit dem Aufbau der Erbsubstanz zusammen, die sowohl im Virus als auch im Wirt dieselben Bausteine benötigt. Das Team um Andreas Dräger ist sich sicher, dass diese Ergebnisse eine wichtige Grundlage für die schnellere Entwicklung von viralen Hemmstoffen darstellen. Die Gruppe will ihre Methoden nun auf weitere Viren anwenden. Erste Hemmstoffe für ihre gefundenen Enzyme sollen in Tiermodellen auf Sicherheit, Toxizität und Wirksamkeit getestet werden.
Die Studie erschien im Fachmagazin „Plos Computational Biology“.
Erhalten Sie jetzt uneingeschränkten Zugriff auf alle interessanten Artikel.
- Online-Zugriff auf das PM-Report Heftarchiv
- Aktuelle News zu Gesundheitspolitik, Pharmamarketing und alle relevanten Themen
- 11 Ausgaben des PM-Report pro Jahr inkl. Specials

